Warszawski Uniwersytet Medyczny jest placówką naukowo-badawczą i ośrodkiem akademickim z ponad 200-letnią historią. Jego kadra to w sumie ponad 2500 nauczycieli akademickich, doktorantów i profesorów, kształcących 10 tys. studentów.
Data science, NLP
AssistMED – asystent badacza do automatycznej analizy danych medycznych w kardiologii
Stworzyliśmy platformę wspierającą badania naukowe oraz algorytmy NLP, które z tekstów medycznych precyzyjnie wyciągają wiarygodną informację na temat chorób, leków i ich dawkowania oraz parametrów echokardiograficznych.
KLIENT

WYZWANIE
Jak wydobyć ze szpitalnej dokumentacji pacjentów cenne informacje, aby wykorzystać je do analiz i badań naukowych? Właśnie ten problem adresuje projekt AssistMED, czyli „Asystent badacza do automatycznej analizy danych medycznych w kardiologii ❤”.
Obecnie większość dokumentacji medycznej przechowywana jest w formie elektronicznej (to jest super), w postaci opisów stworzonych przez lekarzy, w związku z leczeniem pacjentów. Łatwo się domyślić, że czynnik ludzki istotnie wpływa na formę, w jakiej tekst jest sporządzany. Dane nie mają struktury 😨. Jest ich podobno średnio około 80%.
Wyobraź sobie, że każdy lekarz ma swoje przyzwyczajenia, kształcił się gdzie indziej, w innych czasach, używa innego fachowego słownictwa, stosuje inne skróty, działa na innym sprzęcie i … także oczywiście popełnia błędy, czy nie przepada za pisaniną i używa różnych skrótów. Ponadto na rynku są leki o różnych nazwach i zawierające te same substancje czynne, często w innym stężeniu, czy w innej kombinacji.
Uff, czy to już wszystko? No niestety nie, bo z jednej strony w tekstach mamy błędy ludzkie (głównie literówki), a z drugiej bardzo precyzyjne określenia, akronimy, gdzie jedna litera czy kropka zmienia zupełnie znaczenie. Zdarza się język polski i angielski. Ponadto czasem lekarze piszą w różny sposób, że choroba NIE wystąpiła lub nie są jej pewni. Do tego dochodzą dane liczbowe, wyrażone w różnych jednostkach i pochodzące np. z badań echokardiograficznych, które powinny zostać rozpoznane i ustandaryzowane.
Myślę, że wystarczy tych szczegółów i masz już świadomość złożoności zadania! Mieliśmy okazję z sukcesem stawić mu czoła i to było niezwykłą naukową przygodą. 😊 Czasami też bolało.
Jeszcze jedno. Sam projekt został zrealizowany w ramach „Inkubator Innowacyjności 4.0”. Znalazł się wśród zwycięskich projektów w konkursie nr 1 na grant w 2021 roku.
ROZWIĄZANIE
Pewnie się domyślasz, że realizacja wyżej opisanego przedsięwzięcia, to nie wycieczka po sznurku do kłębka.
Jeśli miałbym szukać jakiś obrazowych analogii, to pewnie powiedziałbym, że to była wieloetapowa wyprawa w niedostępne góry ⛰, pełna przygotowań (element badawczo-rozwojowy), w której nie raz wchodziliśmy na tych samych kilka szczytów, w różnych warunkach ❄🌩, przy użyciu innego sprzętu ⛸👡🥾, ciągle starając się poprawić czas wejścia i nie zgubić nic po drodze (precyzja działania)!
Jeśli ciekawi Cię, jak rozwiązuje się takie problemy (bo na przykład czeka Cię coś podobnego, czy też to fajna historyjka do kawy), to czytaj dalej. Teraz będzie konkret, w zwartej formie i w czasie teraźniejszym, aby się łatwiej czytało 😉
Start! Zapoznajemy się z domeną tekstów medycznych i prac naukowych – o co tu w ogóle chodzi? Jaki cel chce tak naprawdę zrealizować nasz klient? Koordynatorem ze strony Uniwersytetu jest lekarz, doktorant i naukowiec Cezary Maciejewski. On ma w głowie trochę gotowych pomysłów, a my przed oczami udostępnioną dużą próbkę opisów lekarskich: rozpoznań chorób u pacjentów ze szpitala, zaleceń leczenia i badań echokardiograficznych. Niezwykłe w tym projekcie są bardzo wysokie kompetencje techniczne Cezarego i znajomość narzędzi z dziedziny data science. Łatwo nam znaleźć wspólny język i zrozumieć wzajemnie swoje myśli. Z ochotą korzystamy z sensownych sugestii i także razem się uczymy.

PLAN DZIAŁAŃ
Plan działań wygląda następująco, przy czym zakładamy, że jego liniowa realizacja odpada i będą cały czas przeploty, a cały projekt jest podzielony na kilka etapów odseparowanych w czasie.
- Wybieramy odpowiednie narzędzia NLP do przetwarzania tekstów i anotacji. Trzeba ustalić, co najlepiej sprawdzi się w projekcie.
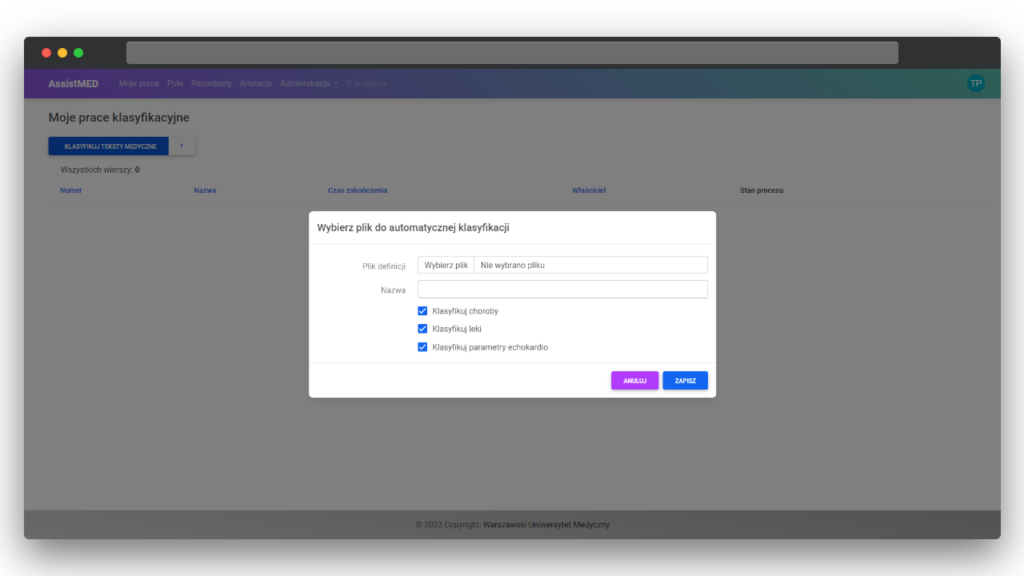
- Projektujemy trzy algorytmy: do wyciągania chorób, leków z dawkowaniem oraz parametrów echokardio, tak aby osiągnąć skuteczność bliską 90%.
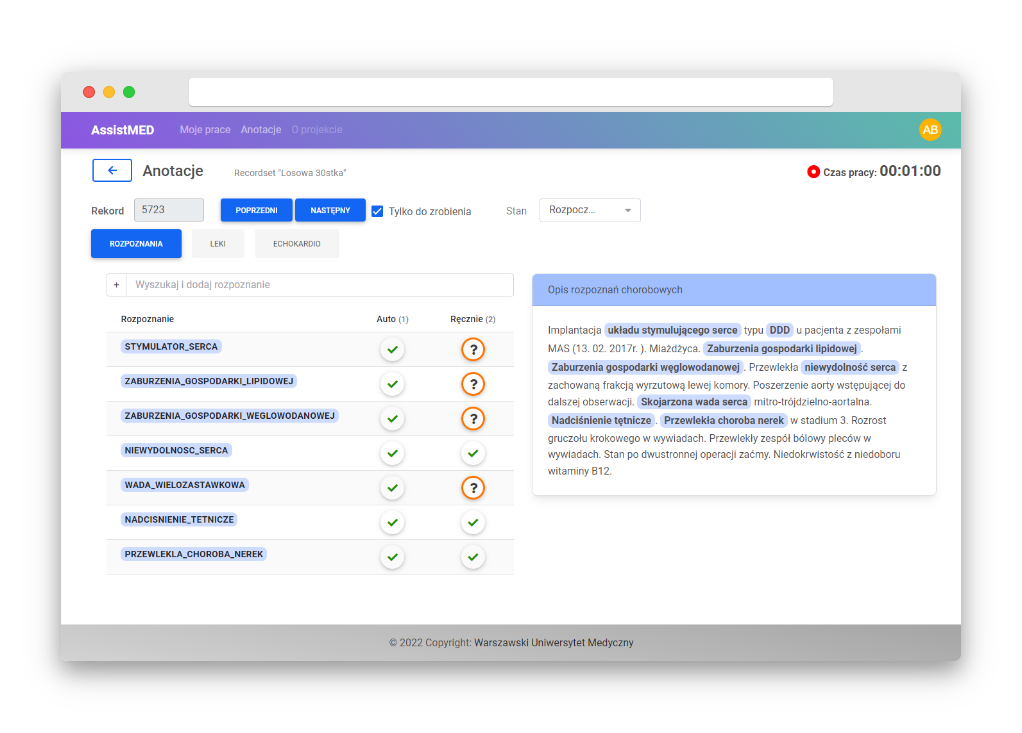
- Przygotowujemy narzędzia do anotacji części danych medycznych (czyli to człowiek rozwiązuje problem, tak aby stworzyć punkt odniesienia dla maszyny).
- Wykonujemy anotacje
- Przechodzimy przez proces ulepszania oprogramowania
- Udostępniamy mechanizmy lekarzom i naukowcom
- Przygotowujemy automatyczne raporty do badań naukowych (obróbka statystyczna)
Etap wyboru narzędzi wiąże się ze zrozumieniem potrzeb. Tu potrzebą jest wysoka precyzja działania i jednocześnie określenie poziomu błędu klasyfikacji, bowiem wyniki mają być zastosowane do wsparcia badań naukowych. Nie można pomachać rękami. Chodzi też o sprawdzenie, jak dobrze człowiek mający wiedzę medyczną, wypada w starciu z oprogramowaniem, w zadaniu strukturalizacji danych. Czy można to bezpiecznie oddelegować maszynie?
System ma być otwarty – tzn. pozwalać na zastosowanie w innych dziedzinach medycyny niż kardiologia, w taki sposób aby końcowy użytkownik – czyli pracownicy naukowi, lekarze, mogli sami użyć go we własnym szpitalu i dostosować. Ba, ma mieć możliwość udostępniania w postaci SaaS (Software as a Service).
Jest rok 2021 (czasy sprzed ChatGPT). Wypracowujemy rozwiązanie z definiowalną bazą wiedzy, przystępną edycyjnie dla osób nietechnicznych, sprytnym autorskim wyszukiwaniem rozmytym fraz (ang. fuzzy search) i wsparciem ze strony gotowych wytrenowanych modeli językowych (machine learning) – dla języka polskiego i MED7 dostępnych poprzez bibliotekę Spacy. Przydają się także stare dobre regexpy.
Oprogramowanie powstaje w Pythonie. Ponieważ operujemy na tekstach w języku polskim, a model MED7 wspierający wykrywanie encji medycznych został wytrenowany na 2 milionach tekstów MIMIC-III, ale po angielsku.. cóż przyda się automatyczna translacja opisów lekarskich z polskiego na angielski. Do tego celu całkiem nieźle sprawdza nam się API Google Translate, przy czym decydujemy się na zastosowanie cache tłumaczeń, aby nie dostać bana od Google za zbyt intensywne użytkowanie…
Tu warto zwrócić uwagę, że budżet projektu nie był duży. Każda oszczędność miała znaczenie. Dbamy w CodiFive o finanse naszych klientów. Ale wracając…
Główne wyzwania wymieniłem na początku. Nie jest prosto jednocześnie pomijać błędy ludzkie w tekście i precyzyjnie rozpoznawać terminologię medyczną. Moje ulubione – „zawał stemi” i „zawał nstemi” 😊 Błędy w końcówkach fleksyjnych słów mają mniejsze znaczenie, ale… nie każde słowo się odmienia.
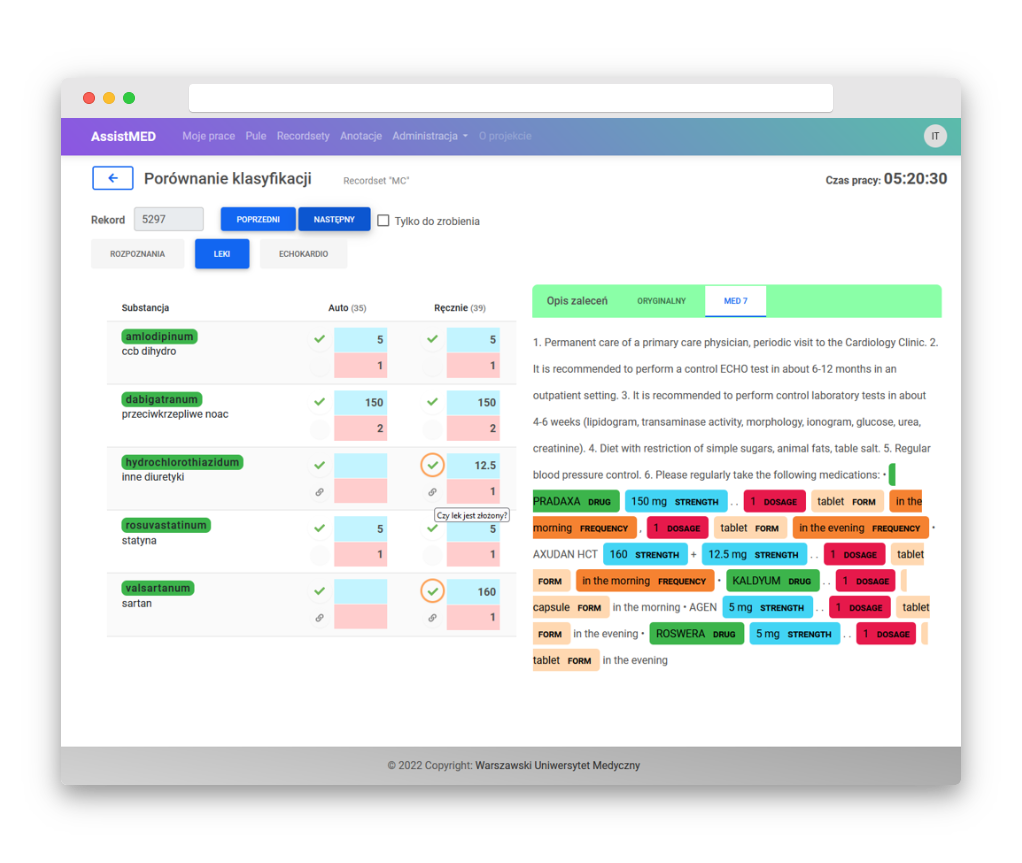
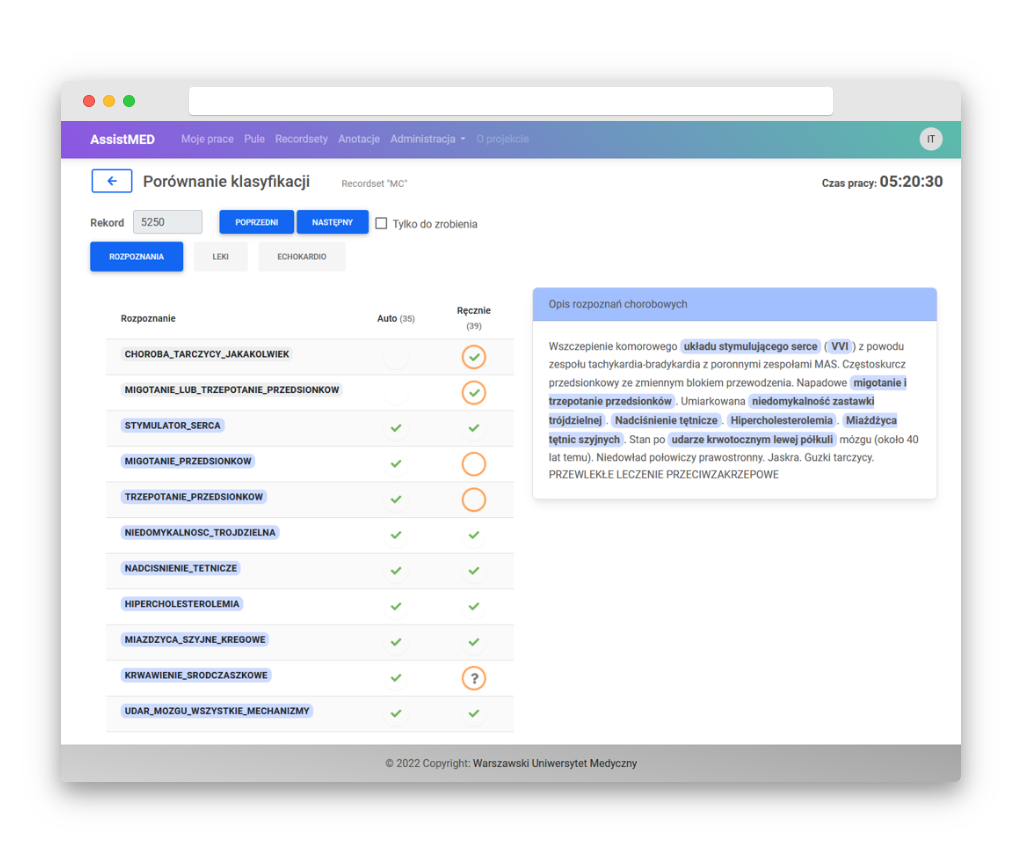
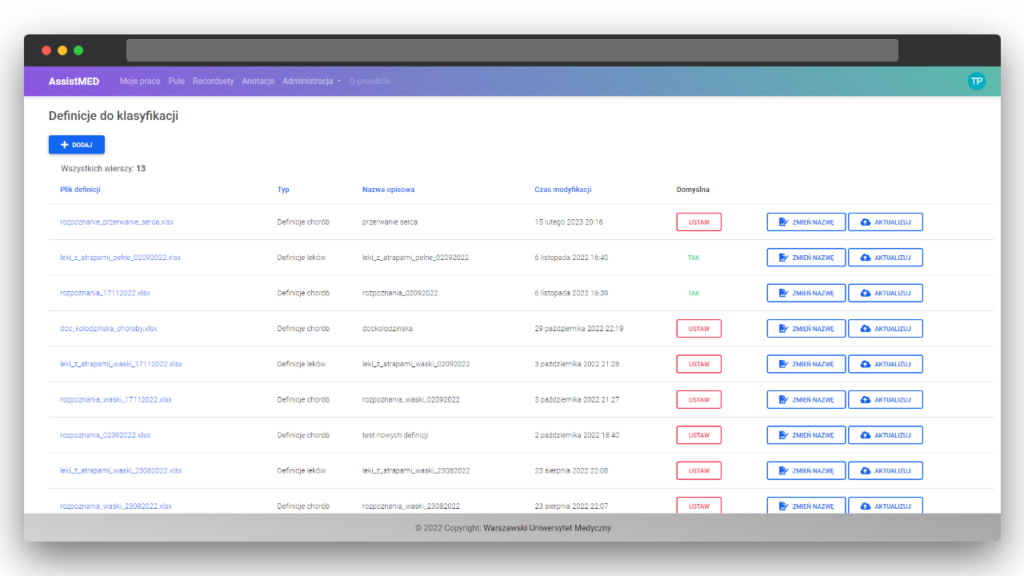
Wyniki działania algorytmów muszą zostać jakoś zwalidowane przez człowieka. Pierwotnie przymierzamy się do narzędzia zwanego Prodigy, ale po próbach okazuje się być niewystarczające z uwagi na ograniczone możliwości definiowania bardziej złożonych anotacji. Także nie zapewnia dobrego UX (User experience), istotnego gdy trzeba przeanalizować prawie 10 tysięcy tekstów znajdując w nich ponad 100 jednostek chorobowych, kilkaset różnych leków i kilkanaście parametrów echokardiograficznych.
W związku z tym tworzymy własne narzędzie do anotacji pozwalające człowiekowi bardzo sprawnie zapisać wszystkie potrzebne informacje i wspierające go na różne sposoby, a także dokonujące pomiaru czasu, jaki musiał poświęcić na analizę tekstów.
Zatrudniamy dwie studentki medycyny, a one pod kierownictwem Cezarego sporządzają potrzebne anotacje. Przygotowane narzędzie umożliwia także porównanie, jak człowiek wypada w konfrontacji z drugim człowiekiem. Daje to możliwość wyciągnięcia dodatkowych wniosków w pracy naukowej.
Ostatnim etapem naszych prac jest przygotowanie raportowania statystycznego. Dla poszczególnych rodzajów klasyfikowanych danych wyliczamy różnego rodzaju miary takie jak czułość, swoistość, PPV, NPV, skuteczność, współczynnik Kappa, Współczynnik Fishera, chi2, macierz błędów, test wilcoxona, mediany itp.
REZULTATY
Innowacyjny system na rynku polskim
Osiągnięcie bardzo wysokiej skuteczności dla języka polskiego 90-100%
Otwartość – łatwa możliwość rozszerzenia na nowe dziedziny medycyny
Projekt zakończył się pełnym sukcesem
Oprogramowanie daje możliwość praktycznego zastosowania w medycynie, badaniach naukowych, badaniach klinicznych leków. Architektura rozwiązania przewiduje skalowanie (większe obciążenie obliczeniami) oraz możliwość wydzielenia modułów, które mogą współpracować z innymi systemami.
23000
125
1194
40
15

Zastosowania
- Analiza dużych zasobów danych medycznych w kardiologii
- Kategoryzacja danych istotna z klinicznego punktu widzenia
- Zwiększenie efektywności prowadzenia w obserwacyjnych badań klinicznych
- Możliwość zastosowania do automatycznych skal ryzyka
Odbiorcy
- Oddziały kliniczne o profilu kardiologiczno-internistycznym
- Lekarze
- Badacze
- Firmy CRO (Clinical Research Organisation) – usprawnienie procesu rekrutacji pacjentów poprzez przyspieszenie prescreeningu i screeningu do badania
- Firmy dostarczające narzędzia informatyczne do zarządzania dokumentacją medyczną
- Podmioty takie jak NFZ czy Narodowy Instytut Zdrowia Publicznego, chcące pozyskiwać dane do analiz epidemiologicznych
Technologie
- Python
- Spacy (NLP)
- MED7, Polish Language Model
- Prodigy
- Django
- Google Translate
- Celery
- RabbitMQ
- Docker
- PostgreSQL
- Linux
Czas trwania
5 miesięcy
Zespół
3 osoby